Am Nationalen Centrum für Tumorerkrankungen Dresden (NCT/UCC) arbeiten Wissenschaftler mit einem modernen Massenspektrometer für die molekulare Pathologie. Mithilfe des Hochleistungs-Apparats lässt sich das Proteinmuster von Geweben bestimmen. Wissenschaftler können hieraus spezifische Merkmale von Tumorgewebe ablesen. Aufgrund seiner Schnelligkeit ist das Gerät für den diagnostischen Einsatz geeignet. Es soll künftig die Tumordiagnostik vor allem bei Patienten des Universitätsklinikums Dresden verbessern und Hinweise auf neue zielgerichtete Therapien geben.
Tumoren lassen sich nicht immer eindeutig histologisch klassifizieren oder vom umliegenden Gewebe abgrenzen. Am NCT/UCC Dresden steht nun eine zusätzliche Bildgebungsmethode zur Verfügung, die die Tumordiagnostik verbessern und Hinweise auf neue zielgerichtete Therapien geben soll. Ein spezielles Massenspektrometer ermöglicht, in kurzer Zeit ein Bild von der lokalen Verteilung der im Tumor und dem umliegenden Gewebe enthaltenen Proteine – dem so genannten Proteom – zu erzeugen. „Mit dem Gerät lassen sich herkömmliche mit Formalin fixierte und in Paraffin eingebettete Gewebeproben innerhalb eines Tages untersuchen. Diese einfache und schnelle Anwendung macht die Methode für den klinischen Alltag relevant“, erklärt Dr. Pia Hönscheid, kommissarische Leiterin des Bereichs Forschung am Institut für Pathologie des Universitätsklinikum Dresden.
Bislang nahm die Analyse von Gewebeproben mittels Massenspektrometrie mehrere Tage in Anspruch. Dadurch war die Methode für die histomorphologische Tumordiagnostik ungeeignet. Das neue Gerät bietet den Pathologen nun die Möglichkeit, herkömmliche Gewebeschnitte und die Proteinverteilung im Gewebe zeitnah zu vergleichen. „Im vergangenen Jahr haben wir damit begonnen, Referenzdaten aus vorhandenen Gewebeproben in das System einzuspeisen. Auf dieser Basis lernt es für bestimmte Krebsarten, Tumorgewebe von anderen Gewebetypen zu unterscheiden. Künftig könnte das Gerät auch unmittelbar Patienten des Universitätsklinikums Dresden sowie ausgewählter Kooperationspartner zu Gute kommen“, sagt Prof. Gustavo Baretton, Mitglied im erweiterten Direktorium des NCT/UCC Dresden und Direktor des Instituts für Pathologie des Universitätsklinikums Dresden.
Mit dem Verfahren können dann frisch entnommene Gewebeproben von Krebspatienten vor allem im Rahmen von Studien analysiert werden. „So gibt es zum Beispiel bei bestimmten Tumoren im Bereich der Gallengangsmündung, den so genannten Ampullenkarzinomen, prognostisch unterschiedliche Subtypen. Wenn wir einen Tumor nicht klar einem der beiden Typen zuordnen können, müssen wir aktuell eine aggressivere Therapie einsetzen. Mithilfe der bildgebenden Massenspektrometrie hoffen wir, künftig auch bei diesen Mischformen eine klare Zuordnung treffen und unseren Patienten die für sie individuell beste und schonendste Therapie anbieten zu können“, sagt PD Dr. Marius Distler, 1. Oberarzt der Klinik und Poliklinik für Viszeral-, Thorax- und Gefäßchirurgie des Universitätsklinikums Dresden.
Neben der Unterscheidung bekannter Tumor-Typen soll die Methode künftig auch neue Tumor-Klassifizierungen ermöglichen und besonders aggressive Bereiche innerhalb von Tumoren sichtbar machen. Ein besonderes Augenmerk der Wissenschaftler gilt seltenen und besonders schwer zu behandelnden Krebsarten, für die bislang kaum Therapieoptionen zur Verfügung stehen. Mit diesem Verfahren können auch Fälle untersucht werden, bei denen nur wenig Tumormaterial vorliegt. „Ein weiterer Vorteil der Methode ist, dass wir im Tumor vorhandene Proteinmuster und perspektivisch auch einzelne Proteine ergebnisoffen abbilden und identifizieren können. Bei herkömmlichen immunhistologischen-Färbungen entscheidet hingegen bereits die Wahl des Antikörper-Panels darüber, welche Proteine sichtbar gemacht werden. Die ergebnisoffene Betrachtung eröffnet die Möglichkeit, neue Zielproteine für die Tumortherapie zu entdecken“, erklärt Prof. Baretton.
Sachsenweit einmaliges molekularpathologisches Verfahren zur Tumordiagnostik
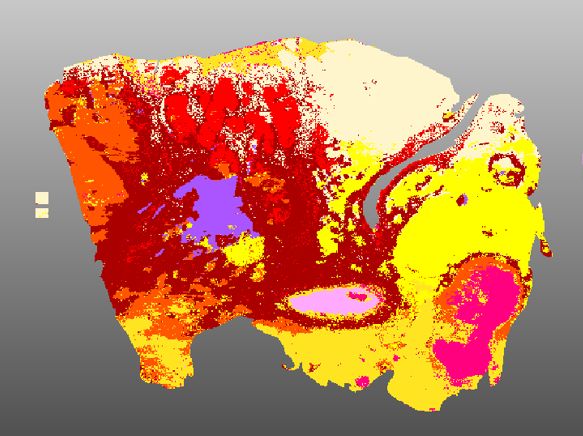
Mithilfe des bildgebenden Massenspektometrie-Verfahrens lässt sich die räumliche Verteilung von Proteinen sichtbar machen. Die orange Färbung zeigt Krebsgewebe eines Tumors der Bauchspeicheldrüse. © NCT/UCC Dresden/Hönscheid
Das NCT/UCC nutzt ein MALDI-TOF-MS-Gerät der Firma Bruker (Modell „Rapiflex Tissuetyper“). Das Kürzel MALDI-TOF-MS steht für „Matrix Assisted Laser Desorption Ionization Time of Flight Mass-Spectrometry“. Bei dieser Methode werden zunächst Protonen aus einer Matrix gleichmäßig auf die zu untersuchende Probe übertragen. Anschließend werden die geladenen Moleküle mittels eines gepulsten Lasers in ein Vakuum-Rohr übertragen und durch eine angelegte Hochspannung beschleunigt. Da alle Ionen zu Beginn die gleiche Bewegungsenergie haben, brauchen sie je nach Masse unterschiedlich lange, bis sie auf einen Detektor treffen. Dieses Masse-Ladungs-Verhältnis lässt sich berechnen, grafisch darstellen und mit konventionell bearbeiteten Gewebeschnitten vergleichen. Bei jeder Probe rastert das System zehntausende Messpunkte ab und erzeugt ein Datenvolumen von bis zu 100 Gigabytes. Deshalb stellt die Data Management and Analytics Unit am NCT/UCC Dresden einen großen Datenspeicher zur Verfügung, in dem Messdaten gesammelt werden können.
Das Gerät wurde aus Mitteln des NCT/UCC Dresden finanziert, die im Verhältnis 90 zu zehn durch den Bund und das Land Sachsen bereitgestellt werden. Der laufende Betrieb wird durch das Universitätsklinikum Dresden, die Medizinische Fakultät der TU Dresden sowie durch Fördermittel finanziert.